알고리즘은 컴퓨터과학의 기초분야이며, 특히 컴퓨터 프로그램을 최적화 하는데 있어서 중요하다. 우리 연구실에서는 암호학, 생명정보학, 검색엔진 분야의 알고리즘을 연구하고 있다. 최근에는 현대의 컴퓨터 구조에 맞춰 멀티코어와 캐시에 최적화된 알 고리즘을 개발하는 연구를 진행하고 있다.
스트링 알고리즘
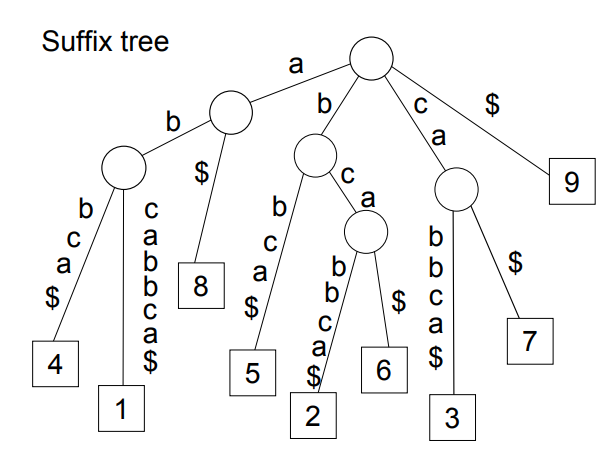
정보를 표현하는 가장 간단하고 자연스러운 방법은 문자열(string)을 이용하는 것이다. 문자로 표현된 데이터를 정렬(sorting), 압축(compression) 및 검색(search)하는 문자열 알고리즘과 full-text index 등 문자열 자료구조에 대한 연구가 활발히 진행되고 있다. 우리는 suffix array에서 longest-common-prefix를 linear-time에 계산하는 알고리즘을 개발하였다 (Annual Symposium on Combinatorial Pattern Matching, 2001, Google 인용지수 491). 한글 문자열에 대한 편집거리를 정의하고 편집거리를 구하는 알고리즘을 개발하였다 (정보과학회 논문지, 2010).
암호학
인터넷 상에서 통신을 할 때, 기밀성 (비밀 유지), 무결성 (데이터 변조 방지), 인증 (통신상대 확인), 그리고 부인방지를 위해서 암호는 필수불가결한 요소이다. 우리는 악의적인 공격에 견딜 수 있는 안전한 시스템을 디자인하는 연구를 진행하였다. 임계암호시스템(Threshold Cryptosystem)에 대한 “공평함”의 개념을 정의함과 동시에 다양한 스킴을 제안하였다 (Int. J. Applied Cryptography 2010). Heterogeneous GPU+CPU system에서 perfect table을 기반으로한 rainbow method의 빠른 parallel implementation을 제안하였다 (Software: Practice and Experience, 2015).
바이오 알고리즘
생명정보학은 전산학과 통계학을 이용하여 생물학적인 문제를 해결하는 분야이다. 우리는 생리학상의 또는 질병의 상태를 나타내는 단백질 샘플들로부터 단백질의 표현상의 차이를 찾아내기 위한 질량데이터 분석 알고리즘을 연구한다. 우리는 Duplex와 triplex mTRAQ 실험에서 peptide 정량을 위한 데이터 분석 알고리즘을 개발하였다 (Journal of Proteome Research 2010, APBC 2011).
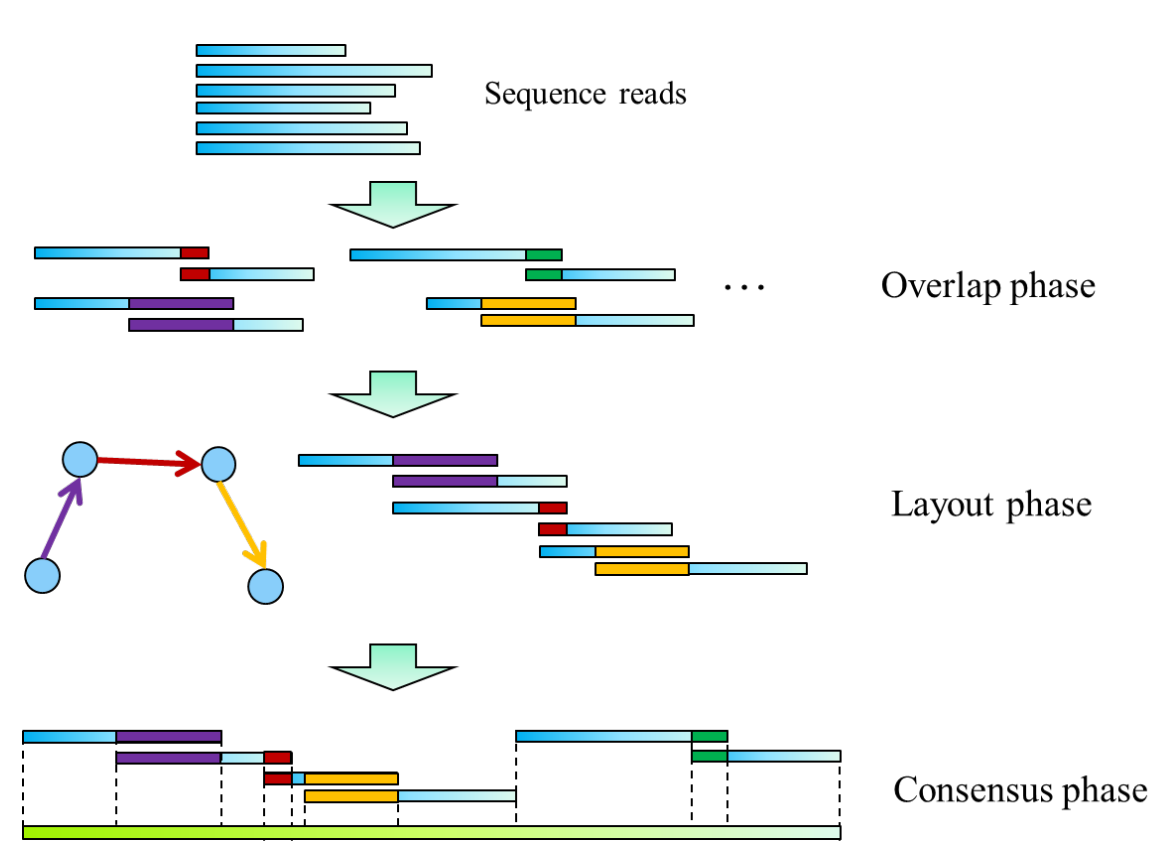
De novo 시퀀스 어셈블리 (De novo sequence assembly)는 레퍼런스 시퀀스 (reference sequence) 없이 주어진 DNA 시퀀스 조각들로부터 하나의 DNA 시퀀스를 재조립하는 작업이다. De novo 시퀀스 어셈블리 작업은 Overlap, Layout, Consensus 단계로 진행되는데 overlap 단계가 가장 많은 시간을 차지한다. 컴퓨터 이론 분야에서는 이 overlap 단계를 All-Pairs Suffix-Prefix 문제로 정의하고 많은 연구가 진행되어왔다. 우리는 All-pairs suffix-prefix 문제를 빠르게 해결하는 알고리즘을 개발하였다 (Theoretical Computer Science, 2017).
그래프 알고리즘
대용량의 그래프 데이터에 대해서 subgraph isomorphism 문제를 푸는 알고리즘을 개발한다.
금융 알고리즘
금융 분야에서 발생하는 여러가지 문제를 해결하는 알고리즘을 개발하고 실제 데이터에 대하여 알고리즘의 성능을 실험한다.